Bien, después de todos estos meses de abandono, causado mayormente por falta de tiempo y neuronas, retomo el blog (a ver si esta vez cuela), ya como bioinformático. Sí, me estaba sacando el máster. Y el Trabajo Final ha sido "durillo". Así que hoy voy a aprovechar para contestar, por este medio, a algo que muchos me han preguntado en la vida real: ¿Qué carajo es la Bioinformática?
El resumen que suelo usar para abuelas es que es usar "brujería de esa de la de Gugel y Facebuk" pero para bichos y humanos.
Si ya queremos adentrarnos un poco más, y entender de qué estamos hablando, podemos decir que en realidad es simplemente Ciencia de Datos (Data Science) aplicada a problemas biológicos. Una de las características de este campo es que, dependiendo de a qué lo apliques, cambia bastante la forma de trabajar. Por tanto, una formación específica es imprescindible.
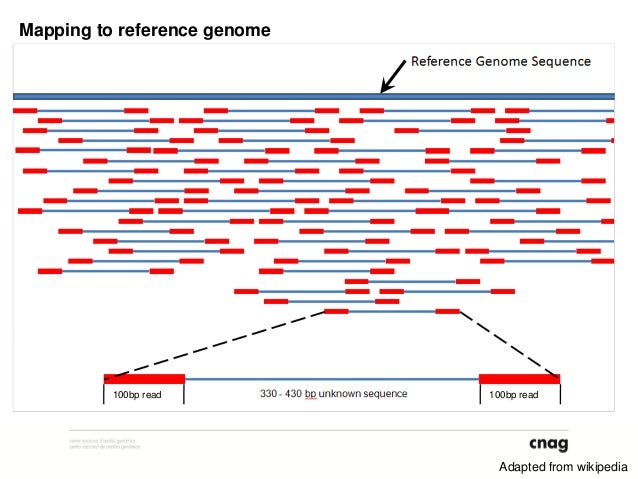 |
| Ejemplo de cómo se superponen los diferentes fragmentos al mapear. |
Este tipo de secuenciación, a pesar de ser más rápida y barata, conlleva una serie de complicaciones técnicas, además de ser muy costosa desde el punto de vista computacional. Por tanto, necesitas a alguien que pueda manejar gran cantidad de datos. Sin embargo, un científico de datos tal cual carece de los conocimientos específicos necesarios para poder trabajar con datos biológicos. Así es como nace la Bioinformática.
Sin embargo, a lo largo de las últimas dos décadas, este campo ha ido creciendo y, aunque todavía está en pañales para muchas cosas, ya cubre todo un conjunto de técnicas y campos, conocidos como "ómicas" (genómica, transcriptómica, proteómica, etc.). Todos estos están más o menos relacionados, pero su principal factor en común es que todos generan gran cantidad de datos, y necesitan potentes y especializadas herramientas matemáticas para poder sacarles provecho.
Hoy en día, con un mercado cada vez más grande de técnicas para obtener estos datos, el campo no deja de expandirse, siendo utilizadas cada vez más en diferentes campos de investigación y ya tienen un pie bien asentado en la práctica médica por su capacidad de diagnóstico de muchas enfermedades hereditarias. Veremos qué más nos traerá este campo en un futuro.

